Viviane Slona (a) Isabelle Glockea (a) Ran Barkai (b), Avi Gopher (b), Israel Hershkovitz (c), Matthias Meyer (a).
a Department of Evolutionary Genetics, Max Planck Institute for Evolutionary Anthropology, Leipzig, Germany
b Department of Archaeology and Ancient Near East Cultures, The Lester and Sally Entin Faculty of Humanities, Tel-Aviv University, Israel
c Department of Anatomy and Anthropology, Sackler Faculty of Medicine, Tel-Aviv University, Israel
Los restos óseos de faunas prehistóricas son a menudo demasiado fragmentaria para permitir la identificación a nivel de especie en base a la morfología, lo que complica los intentos de recuperar las secuencias de ADN antiguo de estos restos. Se diseñó un nuevo enfoque que permite la detección a gran escala de los restos no diagnóstico, para la conservación de ADN mitocondrial antiguo. Después de la extracción de ADN y los preparativos de la biblioteca de ADN, las bibliotecas se enriquecen de ADN mitocondrial utilizando un conjunto de sondas, que cubren la totalidad de los genomas mitocondriales de 242 especies de mamíferos. Se demuestra que la eficiencia de enriquecimiento mitocondrial usando sondas de captura de mamíferos no es sustancialmente menor en comparación con el enriquecimiento utilizando sondas específicas de la especie.
Las excavaciones en la cueva Qesem (Israel), que data de 420-200 ka, han dado un gran conjunto de restos óseos y dentales, incluyendo un pequeño número de dientes de homínidos. A medida que los acontecimientos recientes en la extracción de ADN antiguo, y métodos de preparación de la biblioteca han permitido la generación de los datos de secuenciación de muestras del Pleistoceno medio, que tuvo como objetivo investigar la viabilidad de la recuperación de las moléculas de ADN antiguo de especímenes procedentes de la cueva Qesem. La investigación actual se llevó a cabo utilizando cuarenta y dos restos de fauna de seis diferentes áreas de la cueva, como un estudio preliminar que podría ser utilizado, permanece para su posterior análisis genético. Por desgracia, las bibliotecas de ADN a partir de muestras de Qesem Cave produjeron pocas secuencias que se pueden asignar a un genoma mitocondrial de los mamíferos. Estas secuencias no muestran la estructura del antiguo daño en el ADN que se espera de secuencias endógenas de esa edad. Por lo tanto, no hemos podido detectar la presencia de antiguas moléculas de ADN endógenas en los restos de fauna de la cueva Qesem.
Los recientes acontecimientos en la extracción de ADN antiguo (Dabney et al., 2013) y los métodos de preparación de la biblioteca (Gansauge y Meyer, 2013) han hecho que sea factible recuperar material genético de muestras antiguas fuera de permafrost, ya en el Pleistoceno Medio (Dabney et al., 2013 y Meyer et al., 2014). Sin embargo, la preservación de las moléculas de ADN en el tiempo, tanto en términos de cantidad y calidad, es muy variable, ya que pueden verse afectados, entre otros, por la temperatura del suelo, la acidez, la salinidad, la exposición a la luz solar o la disponibilidad de agua libre y oxígeno ( Lindahl, 1993 Bollongino et al., 2008 y Sawyer et al., 2012). En particular, los climas cálidos parecen afectar negativamente a la conservación de las moléculas de ADN antiguo (Bollongino et al., 2008 y Allentoft et al., 2012). Aunque se necesitan 100 mg o menos de polvo de hueso / diente por técnicas de extracción de ADN actual (Dabney et al., 2013), el muestreo sigue siendo un proceso destructivo. Por lo tanto, la preservación antiguo ADN se determina mejor en los restos de fauna o fragmentos no diagnóstico, antes de probar los fósiles de homínidos, en el supuesto de que los restos óseos de lugares similares en un sitio arqueológico habrían estado expuestos a condiciones ambientales similares.
La detección de muestras antiguas para la preservación de ADN se dirige de forma rutinaria el genoma mitocondrial (MT), un pequeño, circular y no recombinar-molécula de ADN que se hereda por vía materna. La ocurrencia de ADNmt en las copias por célula más altos, en comparación con el ADN nuclear, aumenta las posibilidades de recuperación de ADN antiguo (Pääbo et al., 2004). Además, el rápido ritmo de evolución ADNmt permite la identificación de organismos discretos basados en secuencias mitocondriales, que es particularmente importante cuando se trabaja con fragmentos de hueso o de los dientes de origen incierto. En el pasado, la reacción en cadena de la polimerasa (PCR) se ha utilizado para amplificar tramos cortos de ADN mitocondrial que llevan diferencias de secuencia distintivos entre especies estrechamente relacionadas, por ejemplo en el gen de citocromo b (Loreille et al., 1997 y Burger et al., 2002 ). Sin embargo, este enfoque no es aplicable a una amplia gama de taxones vagamente relacionados. Además, los métodos basados en la PCR no son lo suficientemente sensibles como para detectar los pequeños rastros de ADN altamente degradado que pueden estar presentes en muestras muy antiguas o en los procedentes de sitios bajo condiciones climáticas difíciles (Dabney et al., 2013). Por el contrario, los métodos basados en bibliotecas permiten la amplificación de todos los fragmentos de ADN recuperados en un extracto de ADN. Esto se consigue uniendo engarces de ADN con secuencias conocidas a cada fragmento de ADN antes de la amplificación por PCR. bibliotecas de ADN pueden entonces ser secuenciados directamente en un secuenciador de alto rendimiento, recuperando así las secuencias de fragmentos de ADN al azar. Esta simple estrategia es a veces suficiente para determinar la fuente de especies de una muestra antigua (Blow et al., 2008), pero debido a la dominancia de ADN microbiano en la mayoría de los extractos de ADN antiguo, que tiene un costo prohibitivo para el cribado de grandes conjuntos de muestras. Una alternativa más rentable es enriquecedor bibliotecas de ADN para secuencias mitocondriales. Esto se logra mediante la hibridación de la biblioteca para piezas sintéticas de ADN ( 'cebos') que llevan la secuencia de interés. captura por hibridación ha utilizado con éxito para generar secuencias del genoma del ADNmt, incluso a partir de ADN muy degradado, como de oso y restos de homínido Sima de los Huesos (España) (Dabney et al., 2013 y Meyer et al., 2014). Una limitación de captura de hibridación es que la selección de los cebos de captura apropiadas requiere un conocimiento a priori de la especie en estudio.
Tabla1 Identificación de especies de las muestras estudiadas y características de la biblioteca de ADN generados a partir de cada uno, por área de muestreo. Se describen un enfoque novedoso, "captura de mamíferos mitocondriales ", que permite la detección a gran escala de los restos de fauna y no diagnóstico, para la preservación de ADN antiguo. Para este propósito, utilizamos una piscina cebo que incluye secuencias del genoma de ADNmt de 242 especies de mamíferos, entre ellos la mayoría de las especies (o parientes cercanos de los mismos) que se encuentran en los sitios de mediados y finales del Pleistoceno típicos. Se demuestra la eficacia de la captura de mamíferos mitocondrial de muestras de osos de cuevas ya se sabe que contienen ADN, y el uso de este método para determinar el estado de conservación de ADN en el Pleistoceno Medio restos de la cueva Qesem.
Tabla 2.Lista de las especies de mamíferos incluido en el grupo de sondas de captura de mamíferos mitocondrial.
Qesem Cave es una cámara kárstico situado en un terreno de piedra caliza montañosa a unos 12 kilómetros de la moderna ciudad de Tel-Aviv (Israel). Hominin ocupación de la cueva osciló entre hace 420 y 200 mil años (ka) (Barkai et al., 2003, Gopher y col., 2010 y Mercier et al., 2013), como lo demuestra el descubrimiento de varios dientes de homínidos (Hershkovitz et al., 2011), artefactos líticos pertenecientes a la Acheulo-Yabrudian Complejo Cultural (Barkai y Gopher, 2013), la evidencia para el uso de fuego (Karkanas et al., 2007, Barkai y Gopher, 2013 y Shahack-Gross et al. , 2014), y miles de restos óseos y dentales de fauna (Stiner et al., 2009, Stiner et al., 2011 y Blasco et al., 2014). Los sedimentos en el sitio consisten sobre todo en las capas de color oscuro de arcilla, muy probablemente procedente de la infiltración de terra rossa (Karkanas et al., 2007). La temperatura media anual de la región es de aproximadamente 20 ° C. La precipitación media es de ~ 500 mm por año, a pesar de la precipitación anual puede variar drásticamente.
El análisis morfológico de los restos dentales de homínidos de Qesem cueva revela su similitud con las posteriores Los seres humanos Skhul / Qafzeh temprana anatómicamente modernos, aunque con algunos rasgos neandertales similar (Hershkovitz et al., 2011), dando a luz varios escenarios evolutivos que representan estos rasgos anatómicos . Los datos genéticos de los restos dentales permitirían seguir investigando la identidad de los homínidos Qesem Cave, así como para dilucidar su relación con otros homínidos arcaicos para los que las secuencias de ADN ya han sido generados (por ejemplo, Meyer et al., 2012, Meyer et al., 2014 y Prüfer et al., 2014). La abundancia de restos de fauna en la cueva Qesem ofrece la oportunidad de estudiar la preservación de ADN antiguo en diferentes áreas de la cueva. Este estudio preliminar podría ser utilizado para seleccionar hominin sigue para un análisis genético, disminuyendo así muestreo destructivo de estos fósiles valiosos. Por otra parte, muchos de los restos de fauna de la cueva Qesem son demasiado fragmentarios para permitir la identificación a nivel de especie en base a su morfología (Stiner et al., 2009 y Blasco et al., 2014). La identificación de las especies en función de su material genético ayudaría a echar más luz sobre los patrones del entorno y de la caza de los homínidos de Qesem.
Los restos óseos de los sitios arqueológicos a menudo incluyen una gran proporción de fragmentos no diagnóstico, como es el caso en la cueva Qesem (Stiner et al., 2009 y Blasco et al., 2014). Se demuestra que el enriquecimiento selectivo de secuencias mitocondriales de dos osos de las cavernas antiguas es comparable utilizando sondas específicas de la especie y el uso de la sonda conjunto de los mamíferos. En particular, ya que ambos genomas mitocondriales humanos y neandertales se incluyen en el conjunto de la sonda, que podría ser utilizado para identificar previamente no reconocido de homínidos antiguos restos, siempre que las secuencias interceptado puede autenticado como ADN antiguo endógeno. Por lo tanto, esta sonda conjunto constituye una poderosa herramienta para la detección de restos óseos y dentales sin identificar la presencia de ADN antiguo endógeno. Además, la identificación de especies de restos de fauna no diagnóstico, en función de su material genético podría contribuir a la reconstrucción filogenética de las especies existentes y extintos, así como a los estudios zoo-arqueológica y paleo-ambientales llevadas a cabo en los sitios prehistóricos.
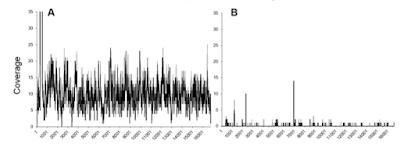
Figura 1.Genoma mitocondrial después del enriquecimiento con sondas específicas osos de cueva (gris), en comparación con el enriquecimiento con las sondas mitocondriales de mamíferos (negro). Además, aunque las sondas de captura mitocondriales de mamíferos son menos eficientes que especies específicas de las sondas en la recuperación de secuencias mitocondriales, especialmente para muestras en las que el ADN es altamente degradado, como el ~430 oso de las cavernas ka de Sima de los Huesos, el conjunto de sonda de mamífero potencialmente puede ser utilizado para capturar secuencias mitocondriales de restos de fauna de una especie de mamífero conocidas. Tal estrategia eliminaría el proceso costoso y requiere mucho tiempo de diseño específico de la especie sondas para cada nueva especie de mamífero estudiados en un marco ADN antiguo; así como ayudar en la verificación de la identificación de especies putativo en los casos de la morfología ambigua. Como experimentos de laboratorio y la secuencia se pueden realizar en paralelo para múltiples muestras de ambas especies conocidos y desconocidos, la captura de mamíferos mitocondrial permite un procesamiento eficiente de un gran número de muestras en el cribado de la antigua preservación de ADN.
Conclusión:
A pesar de utilizar métodos de preparación de extracción de ADN y de biblioteca que se han desarrollado específicamente para hacer frente a las moléculas de ADN antiguo acortados y dañadas (Dabney et al., 2013 y Gansauge y Meyer, 2013), no fuimos capaces de recuperar las secuencias de ADN que podría ser inequívocamente atribuidos a el genoma endógeno de cualquiera de los restos de fauna probadas de Qesem cueva. Sin embargo, el conjunto de sonda de captura de mamíferos mitocondrial, que fue diseñado para el enriquecimiento mitocondrial de especies de mamíferos no identificado, ha demostrado ser una herramienta eficaz para el cribado a gran escala de los conjuntos óseos no diagnóstico, para la preservación de ADN antiguo. El uso de este método para evaluar la conservación del ADN pueden facilitar las decisiones futuras sobre el muestreo.
Referencias:
- Allentoft et al., 2012
- The half-life of DNA in bone: measuring decay kinetics in 158 dated fossils
- Proceedings of the Royal Society B: Biological Sciences, 279 (2012), pp. 4724–4733
- Barkai and Gopher, 2013
- Cultural and biological transformations in the Middle Pleistocene Levant: a view from Qesem Cave, Israel
- ,in: T. Akazawa, Y. Nishiaki, K. Aoki (Eds.), Dynamics of Learning in Neanderthals and Modern Humans, Cultural Perspectives, Replacement of Neanderthals by Modern Humans Series, vol. 1, , Springer, Japan (2013), pp. 115–137
- Barkai et al., 2003
- Uranium series dates from Qesem Cave, Israel, and the end of the Lower Palaeolithic
- Nature, 423 (2003), pp. 977–979
- Blasco et al., 2014
- Subsistence economy and social life: a zooarchaeological view from the 300 kya central hearth at Qesem Cave, Israel
- Journal of Anthropological Archaeology, 35 (2014), pp. 248–268
- Blow et al., 2008
- Identification of ancient remains through genomic sequencing
- Genome Research, 18 (2008), pp. 1347–1353
- Bollongino et al., 2008
- Environment and excavation: pre-lab impacts on ancient DNA analyses
- Comptes Rendus Palevol, 7 (2008), pp. 91–98
- Gansauge and Meyer, 2013
- Single-stranded DNA library preparation for the sequencing of ancient or damaged DNA
- Nature Protocols, 8 (4) (2013), pp. 737–748
- Gansauge and Meyer, 2014
- Selective enrichment of damaged DNA molecules for ancient genome sequencing
- Genome Research, 24 (2014), pp. 1543–1549
- Gilbert et al., 2005
- Assessing ancient DNA studies
- Trends in Ecology and Evolution, 20 (10) (2005), pp. 541–544
- Gopher et al., 2010
- The chronology of the late Lower Paleolithic in the Levant based on U-Th ages of speleothems from Qesem Cave, Israel
- Quaternary Geochronology, 5 (2010), pp. 644–656
- Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructed from ultrashort DNA fragments
- Proceedings of the National Academy of Sciences of the United States of America, 110 (2013), pp. 15758–15763
- Karkanas et al., 2007
- Evidence for habitual use of fire at the end of the Lower Paleolithic: site-formation processes at Qesem Cave, Israel
- Journal of Human Evolution, 53 (2007), pp. 197–212
- Sawyer et al., 2012
- Temporal patterns of nucleotide misincorporations and DNA fragmentation in ancient DNA
- PLoS ONE, 7 (3) (2012), p. e34131
- Stiner et al., 2009
- Cooperative hunting and meat sharing 400–200 kya at Qesem Cave, Israel
- Proceedings of the National Academy of Sciences of the United States of America, 106 (2009), pp. 13207–13212
- Stiner et al., 2011
- Hearth-side socioeconomics, hunting and paleoecology during the late Lower Paleolithic at Qesem Cave, Israel
- Journal of Human Evolution, 60 (2011), pp. 213–233


No hay comentarios:
Publicar un comentario