INTRODUCCION
Debido a la configuración
geológica compleja de la meseta Qinghai-Tíbet (QTP), los efectos de la
orogénesis histórica y oscilaciones climáticas sobre la historia evolutiva de
las plantas en esta región siguen siendo controvertidas. El plan de ahorro,de 2.500.000
kilometros, con una altitud media mayor de 4.000 m, es el más alto y una de las
más extensas mesetas de la Tierra. El
QTP comenzó en el Eoceno temprano, cuando el subcontinente indio chocó con
Eurasia; pero los extensos
levantamientos de la meseta no comenzaron hasta el último Oligoceno. Estos se produjeron al menos cuatro
veces desde a través del Oligoceno Mioceno inferior, Mioceno Medio, Mioceno
superior, Plioceno y Pleistoceno temprano .Además,
la evidencia geológica sugiere que tres o cuatro glaciaciones cuaternarias se
han producido en el QTP. La mayor
glaciación fue la Glaciación Naynayxungla
Como región topográficamente
compleja, contiene 9.000 a 12.000 especies de plantas vasculares de 1.500 géneros. Al menos 20% de estas especies y 50 géneros son endémicos. El origen y la distribución actual de
estos taxones endémicos son el producto de la interacción continua entre los
procesos evolutivos y los cambios ambientales en la región. Las historias evolutivas de la mayoría
de la flora pueden dividirse a grandes rasgos en tres etapas. Durante la primera etapa (Eoceno al
Mioceno Temprano) comienza el levantamiento y se extiende hacia el sur, y
muchos géneros o linajes con alta diversidad de especies en el QTP se
divergieron de sus especies hermanas. En la segunda etapa (Mioceno Medio a
Plioceno), se producen varios
levantamientos extensos que dan lugar a las cordilleras de esta región. Estos eventos, crearon la
fragmentación del hábitat intenso y activaron la diversificación de muchos
géneros. Parece que la mayor
parte de la diversidad de especies endémicas en esta región se originaron
durante este período. Desde el
Pleistoceno hasta la actualidad (la tercera etapa), el QTP ha sufrido
oscilaciones climáticas del Cuaternario. Sin
embargo, la magnitud de los cambios climáticos fueron heterogéneos en
diferentes partes debido a su compleja topología. Aunque algunas especies fueron
llevadas a la extinción, la mayoría de las especies experimentaron una
demografía dinámica. Otras
especies pueden haber experimentado divergencia intraespecífica. La mayoría de la investigación se ha
centrado en las dos últimas etapas. Por
el contrario, los recientes estudios se han producido en la última etapa.
Hasta ahora, ha habido muy poca
investigación sobre la correlación entre eventos orogénicos y evolutivos en las
tres etapas en una sola especie, Juniperus microsperma (WC Cheng y LK Fu) RP Adams nos ofrece
un excelente modelo para abordar este tema. Juniperus L. es
un elemento importante de los ecosistemas áridos y semiáridos en todo el
hemisferio norte , pero J . microsperma es una conífera extremadamente rara
que actualmente solo se conoce desde el Valle Parlung Zangbo, al sureste de esta
zona . En estudios anteriores, se encontró J. microsperma en clados
filogenéticos que contienen J. semiglobosa , J. sabina y enebros norteamericanos de hoja lisa. Juniperus microsperma parece haber divergido de las especies
estrechamente relacionadas con el
Mioceno temprano. Aunque un grupo de J . microsperma
con un grupo de especies alopátricas que crecen lejos, comparte rasgos
morfológicos y los componentes de la hoja terpenoides con dos especies
simpátricas, J . convallium y J . saltuaria , indican posible flujo de genes de J . microsperma a las dos últimas especies o viceversa.Teniendo
en cuenta la actual distribución estrecha y de pequeño tamaño de la población
de J .microsperma , y la distribución mucho más amplia y
de mayor tamaño de la población de su especie hermana, J. sabina y J. semiglobosa , es altamente probable que este raro
enebro experimentó una historia demográfica dinámica. Estas hipótesis merecen mayor
validación .
En este estudio, han empleado
loci nucleares múltiples y dos marcadores cpDNA para investigar la variación
genética entre las poblaciones de J. microsperma y sus congéneres, J.semiglobosa , J. sabina , J. convallium , J. saltuaria y J. tibetica . Luego,
mediante la utilización de una serie de enfoques genéticos filogenéticos y de
la población. La historia de la especiación de J. microsperma y sus parientes cercanos, así como su
historia demográfica se infiere sobre la base de la variación genética entre y
dentro de las especies.
MÉTODOS
El muestreo y recogida de datos
A lo largo de la distribución natural conocida de J. microsperma en el QTP, muestras de hojas se obtuvieron de siete poblaciones naturales, y cuatro y 11 poblaciones representativas de J. sabina y J. semiglobosa también se recogieron. Además, también compilado un conjunto de datos de secuencias de ADN de 19, siete y ocho poblaciones de J. tibetica , J. convallium y J. saltuaria en el QTP. Las hojas frescas se tomaron muestras y se secaron inmediatamente usando gel de sílice. La latitud, longitud y altitud de cada sitio de muestreo se midió por Extrex SIG.
Análisis filogenéticos y reconstrucción de árboles de especies
Para ambos haplotipos cpDNA y genotipos nucleares, las relaciones filogenéticas entre se establecieron utilizando RED.Anteriormente, se había llevado a cabo una encuesta de población para la variación cpDNA de J. convallium , J. saltuaria y J. tibetica en un tamaño de muestra grande, se aprobaron cinco cloroplasto haplotipos frecuentes para representar estas tres especies. Se utilizó MrBayes 3.2.1 45 para construir el árbol filogenético de los haplotipos cpDNA. Además, se realizó un análisis de componentes principales de los polimorfismos genéticos.
Pruebas de diversidad genética y de neutralidad
Las secuencias nucleares con picos aditivos fueron eliminados y se separaron en dos secuencias de alelos por FASE en el paquete de software DnaSP. Entonces, hemos utilizado las secuencias nucleares para estimar los parámetros básicos de genética de poblaciones, incluyendo el número de sitios segregantes , el parámetro de Watterson , la diversidad de nucleótidos, el número mínimo de eventos recombinantes, número de haplotipo y la diversidad en el mismo paquete de software . Clasificamos los sitios segregantes en cuatro categorías.
Estructura de la población y delimitación genética
Para cuantificar la extensión de la estructura genética de la población, primero calculamos el índice de Wright fijación ( F ST ) y la secuencia de divergencia neta ( D una ) en cada locus ADN nuclear para cada par de especies usando un análisis de varianza molecular tal como se aplica en ARLEQUIN y DnaSP. Del mismo modo, para evaluar la diferenciación de la población dentro y entre las especies, Φ ST a nivel multi-locus se calculó para cada par de especies. Φ ST valores fueron estimados por AMOVA utilizando ARLEQUIN sobre una base locus por locus, así como un promedio a través de todos los loci. La importancia de F ST y Φ ST fueron probados con base en 10.000 permutaciones.
RESULTADOS
La variación genética y la divergencia interespecífica
Se analizaron los datos de secuencia de ADN de ocho regiones nucleares y dos fragmentos de cloroplastos de 239 individuos de 56 poblaciones de J. microsperma (60 individuos de siete poblaciones) y cinco de sus congéneres, J . sabina , J . semiglobosa , J . convallium , J . saltuaria y J . tibetica ( Tabla S1).Entre ellas, las secuencias de ADN de 83 individuos pertenecientes a las antiguas tres especies fueron generadas aquí , y las otras secuencias se recogieron previamente.
Dos fragmentos cpDNA se secuenciaron a través de todos los individuos de J . microsperma , J .Sabina y J . semiglobosa , y se obtuvieron 1504 pb de datos de la secuencia alineados. Cinco sustituciones y dos indeles fueron detectados ( Tabla S2 ), y en estos polimorfismos se identificó un único haplotipo en J . microsperma , y dos haplotipos uno para cada especies; sin haplotipo fue compartido entre estas tres especies. También construimos una red haplotipo cloroplasto, que mostraron que los haplotipos de cloroplastos de las seis especies se agruparon en dos grupos distintos ( Fig. 1 ).
Figura 1.(A)Distribuciones geográficas , ( B )redes y ( C )el árbol de máxima verosimilitud de los 10 clorotipos que se identificaron en Juniperus microsperma , J. sabina , J. y J. semiglobosa tibetica compleja . En (A) y (B), cada círculo representa una población , el color del círculo esquema representa el atributo especies de cada población. En (C), los valores de arranque por encima de 0,5 se muestran en la rama junto a cada nodo .
La secuenciación de los ocho
loci nucleares resultó en 4.504 pb de la secuencia de datos alineados , y 51 ,
58 , 43 , 40 , 62 , y 47 sitios segregantes fueron identificados en J.
microsperma , J. sabina , J. semiglobosa , J. convallium , J . saltuaria y J.
tibetica , respectivamente. La secuencia de diversidad ADN Nuclear se detectó
en todos los loci de cada una de las seis especies, aunque la cantidad de
polimorfismo de nucleótido varió mucho entre loci. Teniendo en cuenta la
diversidad de nucleótidos en silencio y totales como promedio durante ocho loci
, la diversidad de nucleótidos de J. microsperma es moderada entre los seis enebros (Fig 2 )
Figura 2. Las barras representan los valores de la mediana, las cajas representan los rangos intercuartil y bigotes se extienden a 1,5 veces de cada rango intercuartílico.
La significativa diferenciación
de la población y la secuencia de divergencia neta entre las especies fueron
detectadas en casi todos los loci de ADN nuclearlos valores de la diferenciación
mostraron que la diferenciación genética entre J. microsperma y las otras cinco
especies fueron significativas en siete de los ocho loci. Resultados para una
divergencia neta eran similares a los anteriores. Las divergencias estimadas
entre J. microsperma y J. sabina, y entre J. microsperma y J.
semiglobosa, eran comparativamente más bajos que entre J. microsperma y cada uno de J.
convallium , J. saltuaria y J. tibetica. La mediana de unirse a las
redes genotipo construidos para cada locus nuclear ( Fig. 3 ) también mostraron
que J. microsperma está más relacionada con J. sabina y J.semiglobosa
.
Figura 3. Redes para todos los haplotipos detectados en cada uno de los ocho loci nucleares. Cada círculo representa un haplotipo, y cada color representa una especie; el tamaño de cada círculo es proporcional al tamaño de la muestra de cada población. Los círculos negros representan haplotipos que faltan, y los pasos mutacionales son las barras negras.
La mediana también mostró que J. microsperma está más estrechamente relacionado con J. sabina y J. semiglobosa . Como se muestra en la Fig. 3 , genotipos nucleares generalmente se dividen en dos grupos: un grupo está formado por genotipos de J. microsperma , J. sabina y J. semiglobosa , mientras que otro grupo incluye genotipos de J. convallium , J. saltuaria y J. tibetica . La trama PCA con datos idénticos genera un patrón, en la que los individuos del primer grupo del anterior patrón es igual , y las tres especies restantes se agruparon en tres grupos distintos ( Fig. 4 ).
Figura 4. Patrón PCA
Además, el algoritmo de agrupamiento Bayesiano reveló que el número más probable de racimos fue K = 2 cuando se utiliza ocho loci nucleares ( Fig. 5A ). Para K = 2, estarian los dos grupos anteriores; para K = 3, J. microsperma convirtió en un clúster independiente; y para K = 4, individuos de J. convallium , J. saltuaria y J. tibetica se asignaron más de dos grupos. Se ha propuesto que la estructura genética intraespecífica es uno de los factores que juegan un papel en la generación de falsas señales. Por lo tanto, se realizó un análisis posterior de agrupación bayesiano para J. microsperma pero no se encontró ninguna estructura de la población detectable en esta especie.
Empleamos BESTIA para estimar una especie de árboles utilizando un marco bayesiano basado en loci nucleares múltiples para muchos individuos. Lo que proporcionó la agrupación J. microsperma , J. semiglobosa y J. Sabina , y otra agrupación J. convallium , J. saltuaria y J. tibetica , así como para agrupar estos dos clados; además, la relación entre la hermana J. semiglobosa y J. sabina recibió un apoyo moderado ( Fig. 6 ).
Figura 5. La agrupación bayesiano para las personas de los seis enebros basado en ocho loci nucleares; dos, tres y cuatro grupos ( K = 2, 3, 4)
Inferencia de las especies de árboles
Empleamos BESTIA para estimar una especie de árboles utilizando un marco bayesiano basado en loci nucleares múltiples para muchos individuos. Lo que proporcionó la agrupación J. microsperma , J. semiglobosa y J. Sabina , y otra agrupación J. convallium , J. saltuaria y J. tibetica , así como para agrupar estos dos clados; además, la relación entre la hermana J. semiglobosa y J. sabina recibió un apoyo moderado ( Fig. 6 ).
Figura 6.El árbol estaba arraigado con J. communis según estudios filogenéticos anteriores. Las probabilidades posteriores son reportados por encima de los nodos
Pruebas neutrales y demográficos
Se calculó un conjunto de índices para detectar desviaciones del modelo estándar neutral de la evolución molecular en cada locus nuclear. Entre J. microsperma y sus dos especies más estrechamente relacionadas, la media fue negativa de J. microsperma , la media fueron negativas de J. sabina , y la media fueron negativas de J. semiglobosa . A continuación, se realizó la prueba HKA para probar la salida de la neutralidad en el locus individual. Las seis especies fueron probados a su vez con J. communis y nos encontramos sin desviación significativa del modelo neutral. Además, la prueba MFDM indicó que había una probabilidad significativa de selección en el CC1333 locus en J. microsperma . Además, en CC1333 de J. semiglobosa , los resultados indicaron cerca de importancia para la selección. Finalmente, el análisis LAMARC sugirió que J. microsperma ha experimentado grandes cambios en el tamaño de la población.
Modelado aislamiento y la migración entre las especies
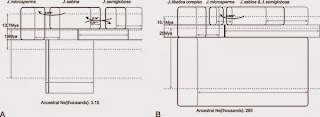
Se realizó en dos poblaciones una simulación IM para estimar parámetros demográficos: el tamaño efectivo de la población, el tiempo de divergencia y la tasa de migración. Los resultados mostraron una estimación de 26,900-45,900 para el tamaño efectivo de la población de J. microsperma . El tiempo de divergencia media se estima en 14 a 21,6 Mya entre J. microsperma y el J. convallium - J. saltuaria - J. tibetica, 13,8 Mya entre J. microsperma y J. sabina y 4,45 Mya entre J. microsperma y J. semiglobosa . Sin embargo, la tasa de migración entre J. microsperma y la otra especie de cinco enebros fue de aproximadamente cero, lo que sugiere que el flujo de genes fue muy bajo. También se realizó una simulación de mensajería instantánea de J. microsperma y sus dos especies más relacionadas; una migración significativa pero menor de J. sabina a J. microsperma , y la migración significativa y bidireccional entre J. semiglobosa y J. Sabina se detectaron ( Fig 7A.); y se estimó que J. microsperma y el MRCA de sus dos especies relacionadas pueden haber divergido 19 Mya. Los resultados mostraron que J. microsperma y sus dos especies más estrechamente relacionadas podrían haber divergido con el MRCA de J. convallium , J. saltuaria y J. tibetica ( Fig. 7B ) a 25 Mya, y J. microsperma divergió de sus dos parientes más cercanos en ca. 16,1 Mya; flujo de genes importante, pero de menor importancia se detectó a partir de sus más cercanos a J. microsperma ( Fig. 7B ).
Figura 7.( A ) Historias para las poblaciones de Juniperus microsperma, J. sabina y J. semiglobosa , y ( B ) las historias de las poblaciones de Juniperus microsperma , J. sabina plus J. semiglobosa y el J . tibetica complejos
DISCUSIÓN
La encuesta genética de la población reveló que la historia evolutiva de J. microsperma se caracteriza por dos acontecimientos cruciales. En primer lugar, la divergencia de J. microsperma en sus dos especies más estrechamente relacionadas, J. sabina y J. semiglobosa , durante el Mioceno temprano, y con el apoyo de que J. microsperma fue aislado tras los primeros levantamientos de la QTP. En segundo lugar,que J. microsperma experimentó una disminucion de población drástica durante el Cuaternario tardío. La especiación y las historias demográficas de J.microsperma proporcionan otro caso en el que la historia evolutiva de las especies relictas en el QTP están estrechamente relacionados con los cambios geológicos y climáticos en esta región sensible .
Las inferencias de árboles de especies bayesiano generaron una topología de buen apoyo que sugería que J. microsperma es hermana del ancestro común de J. semiglobosa y J. sabina; simultáneamente, J. convallium , J . saltuaria y J . tibetica forman otro grupo
El patrón de distribución actual de estas especies y los tiempos de división entre ellos sugirió que la divergencia entre J. microsperma y el antepasado común de sus dos especies más estrechamente relacionadas fue muy probablemente causada por la formación de barreras geológicas provocadas por principios de levantamiento QTP. Aunque J. microsperma sólo se conoce desde el condado de Bomi, en el sureste de QTP a lo largo del Valle Parlung Zangbo ,J. semiglobosa se produce a lo largo de la frontera noroccidental de China y la parte oriental de Asia Central ; y J. sabina es generalizada en el norte de China, Mongolia, del Lejano Oriente de Rusia, Asia Central y Europa del Juniperus semiglobosa y J. sabina tienen distribuciones se superponen en Kirguistán, Tayikistán y la parte noroeste de la mayor parte de China, pero la distribución de J. microsperma está lejos de estas dos especies .
En resumen, el estudio es un estudio de caso único que se centró en las pruebas de la correlación entre la historia evolutiva de una especie antigua y los cambios ambientales históricos en el QTP, y nuestros hallazgos sugiere que QTP eleva y los cambios climáticos del Cuaternario puede haber en forma fundamentalmente la historia evolutiva de las plantas en esta región.
REFERENCIAS
Articulo:Evolutionary origin and demographic history of an ancient conifer (Juniperus microsperma) in the Qinghai-Tibetan Plateau ir a la pagina
- Junipers of the World: The genus Juniperus. 4th edn Trafford Publishing 2014).
- Approximate Bayesian computation in evolution and ecology. Annual Review of Ecology, Evolution, and Systematics 41, 379–406 (2010).
-, , , & Diversification and biogeography of Juniperus (Cupressaceae): variable diversification rates and multiple intercontinental dispersals. New Phytol. 188, 254–272 (2010).
, , , & Evolutionary diversifications of plants on the Qinghai-Tibetan Plateau. Frontiers in genetics 5, 4 (2014).
-, & The relationship between climate change and Quaternary glacial cycles on the Qinghai-Tibetan Plateau: review and speculation.Quaternary International 97-98, 93–101 (2002).







2 comentarios:
¡Hay multitud de nombres de especies sin cursivas! Arréglalo.
Para quien no se percate, el artículo de donde sale todo esto es:
Shang, H.-Y. et al. (2015) Evolutionary origin and demographic history of an ancient conifer (Juniperus microsperma) in the Qinghai-Tibetan Plateau. Scientific Reports, 5, 10216 doi:10.1038/srep10216
http://www.nature.com/srep/2015/150515/srep10216/full/srep10216.html
Al no mencionarlo, tu entrada se puede considerar un plagio, ya que cuentas todo como si lo hubieras hecho tú ("hemos utilizado", "empleamos", "nuestros hallazgos").
Hola Manuel, muchas gracias por avisarme! Creo que los dos problemas mencionados están solucionados.
Buenos dias!!
Publicar un comentario